ゲノム解析の解像度とは
ゲノム解析の解像度とは、検査の方法を理解するのに重要です。ゲノム解析と言っても塩基一つから染色体1本レベルまであるからです。たとえば染色体分染法で代表的なGバンドのバンド一つの大きさが大体5~15Mb というように検出したいものの大きさで検査を選ぶ必要があります。
ゲノム解析の解像度とは、ゲノム配列をどの程度の詳細かつ精確に解析できるかを示す指標です。解像度が高いほど、より小さな遺伝的変異や微細な配列の違いを検出する能力が向上します。この解像度は、使用するシーケンシング技術、サンプリング手法、データ処理のアプローチによって異なります。
● ゲノム解析の解像度を決定する要因
1. シーケンシング技術:
– 次世代シーケンシング (NGS): 高スループットで低コストなNGS技術は、ゲノム全体を迅速に解析することができますが、リード(読み取り断片)の長さが短いため、特定の配列の解像度は限られることがあります。
– 第三世代シーケンシング: シングルモレキュールリアルタイム (SMRT) シーケンシングやナノポアシーケンシングなどは、より長いリードを生成し、高い解像度で複雑な領域や繰り返し配列を解析できます。
2. サンプリング戦略:
– 全ゲノムシーケンシング: ゲノム全体を解析することで、全体的な高解像度のデータを提供しますが、コストが高くなる可能性があります。
– エクソームシーケンシング: タンパク質コーディング領域(エクソン)のみを対象とし、これらの領域に限定した高解像度のデータを提供します。
3. データ解析とインフォマティクスツール:
– シーケンスデータを処理し解析するアルゴリズムやソフトウェアの精度も解像度に影響を与えます。高度なアルゴリズムは、より少ないエラーと高い精度でデータを解読することができます。
4. 参照ゲノムとの比較:
– 解析されたゲノムデータを既知の参照ゲノムと比較することで、変異や違いを特定します。参照ゲノムの質が高ければ高いほど、解析の解像度も向上します。
● 解像度の影響
ゲノム解析の解像度が高いほど、微細な遺伝的変異、点突然変異、小規模な挿入や欠失、そして疾患関連の複雑な遺伝子変異が検出可能になります。これにより、個別化医療、疾患予防、および新しい治療法の開発に役立つ詳細な遺伝情報を提供することができます。高解像度ゲノム解析は、科学研究だけでなく、臨床診断と治療においても重要な役割を果たしています。
染色体検査
染色体検査に使用する細胞は、培養して増殖できるものでなければなりません。
なぜなら、染色体は通常は見えないのですが、細胞分裂するときにだけ見えるようになるからです。
このため、大体は血液を採取して白血球、特にTリンパ球を用います。
まず、末梢血を採取して白血球を集めます。
次に培養液を加えて分裂させるように刺激します。
通常はphytohemaggulutinin(PHA)を添加します。
数日後に分裂している細胞の紡錘体の働きを阻害するために、コルセミドなどの薬剤をを添加して
分裂中期で細胞分裂を停止させます。
関連記事:紡錘体
細胞を低張液で処理して染色体を分散させた後てから染色体を、固定しスライドグラスに広げて診断法に応じていくつかある染色方法のなかから必要なものを実行します。
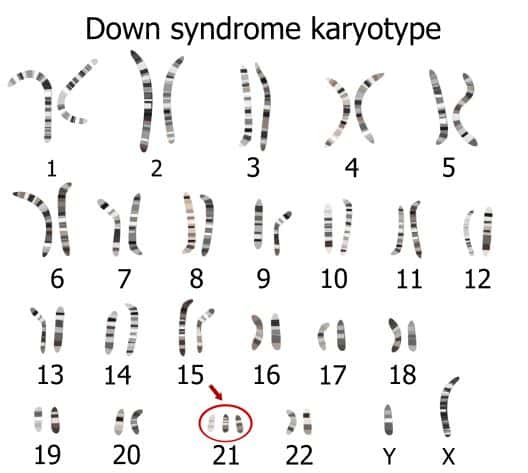
染色体の1~22番と性染色体1本のセットをハプロイド(一倍体)といいますが、これには30億対の塩基が含まれています。
解像度
どのくらいの小ささまで見えるのか?ということです。
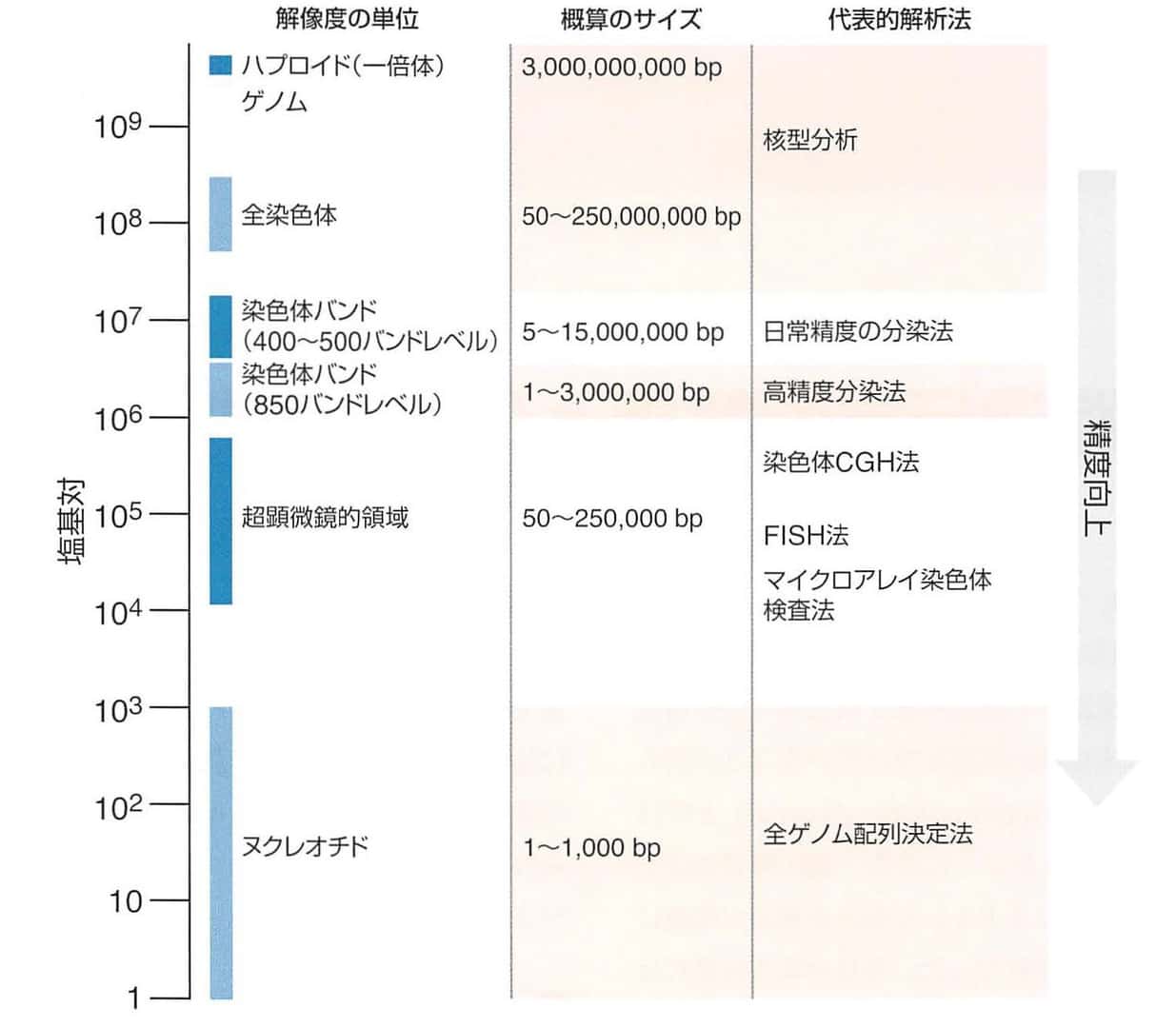
こちらの図はトンプソン&トンプソン遺伝医学第2版から引用しました。
Gバンド
たとえば、ハプロイドをみるなら普通の染色方法で十分ですね。
染色体1本を見るにも普通の染色(Gバンド)で十分です。
5~15Mb(mega base = 100万塩基)くらいまでこの普通の分染法で十分です。
高精度分染法
これに対して、Gバンドのバンドより小さいものは大きな欠失や重複などはGバンドで可能ですが、それより小さなものは目を凝らしても見えませんので、もっと小さいものをみえるような解析法を取ります。
これが高精度分染法と呼ばれるもので、1~3Mbを見ることができます。
1980年代初頭に高精度分染法が開発され、2~3Mb程度の大きさの微細なゲノムの欠失や重複により引き起こされる微細欠失症候群(microdeletionsyndrome) がいくつも発見されることになりました。
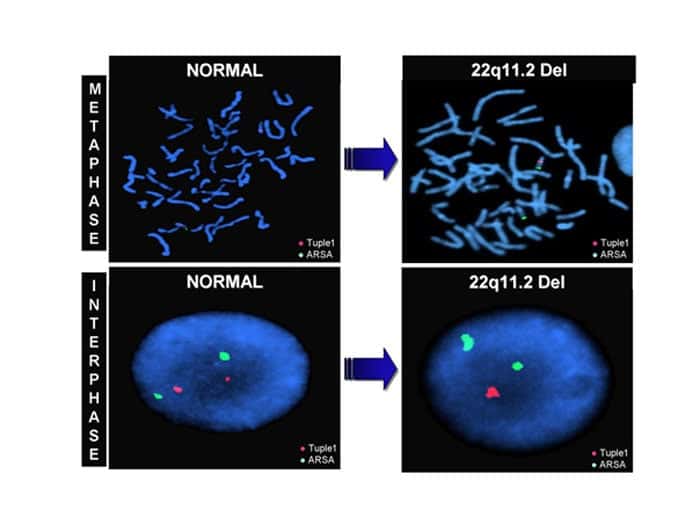
FISH法
特定領域を詳細に解析する高精度分染法は1990年代初頭に蛍光insituハイブリダイゼーション(fluorescence in situ hybridization : FlSH)法が開発されたことで置き換わりました。FISH法は特定のDNA配列の有無を検出したり、染色体や染色体領域の数やその構成を細胞内の"元の位置(in situ)" で評価する方法です。
25~50Kbを検出することができます。
マイクロアレイ
さらに小さな10Kb~ を検出できます。
これに関しては別に説明します。
全ゲノムシークエンス
シークエンサーという機械を使って、すべての塩基配列を決定するものです。
1~1000baseの検査に対応しています。
大きな欠失や重複の検出には向いていません。









