次世代シークエンサー next generation sequencer NGS
次世代シークエンサー next generation sequencer NGSとは?
シークエンシングsequencingは、連続したもの、その順序、という意味を持つ英語です。
わたしたちが目で文字をスキャンして文章を読むのと同じように、「遺伝の書」ともいえるDNAの「文字」である塩基配列(シークエンス)を「読み取る」のがシークエンシングです。シークエンシングについてはリンク先のべページをご覧ください。
シークエンシングする機械のことをシークエンサーと呼びます。
マイクロアレイ法はオリゴヌクレオチドと言う短いDNA断片をチップに貼り付けて、それと反応するDNA領域があるかどうかを見る、というものですが、マイクロアレイとは対照的に、シークエンサーでは、与えられたDNAやcDNA分子の核酸配列を直接決定します。
DNAシークエンシングへの最初の大きな取り組みは、ヒトゲノムプロジェクトである。このプロジェクトは、サンガー塩基配列決定法(鎖終結法)と呼ばれる第一世代の塩基配列決定法を用いたもので、たった一人のDNA塩基配列を決定するのに13年の歳月と世界中のコンピューターとマンパワーと30億ドルの費用をかけ、2003年に完成しました。
これに対して2000年代後半から出てきた次世代シークエンシングは、キャピラリー電気泳動を用いた従来のサンガーシーケンシングと比較して、大量並列のシーケンシングを一気に行うことで、シーケンシング能力に革命をもたらし、桁違いのデータを低コストで提供するということで、第二世代シーケンシング法とか次z世代シーケンシング(NGS)とかと呼ばれる根本的に異なるシークエンシングアプローです。
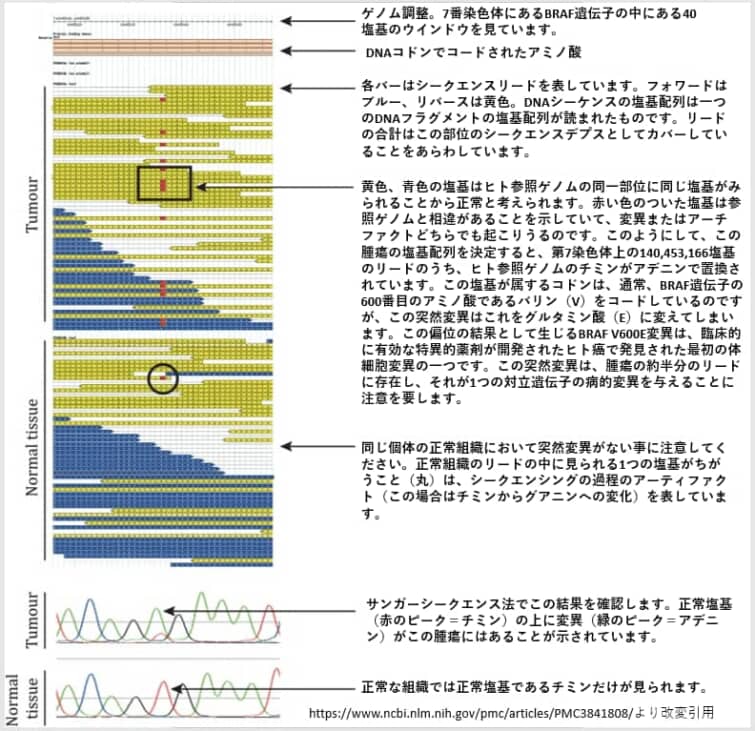
次世代シーケンシング(NGS)、大規模並列シーケンシング、ディープシーケンシングは、こうしたゲノム研究に革命をもたらしたDNAシーケンシング技術を表す同義語です。この技術を使ってシークエンスする機械を次世代シークエンサーといいます。NGSを使用すると、ヒトゲノム全体の塩基配列を1日以内に決定することができます。ゲノム研究においては、NGSは従来のサンガーシークエンシングにほぼ取って代わられてきていますが、ルーチンの臨床現場にはまだ導入されていません。NGSで異常が検出された部分はサンガー法で確認をしています。
しかし、サンガー法だと異常を検出するのに時間がかかるため、おおざっぱにNGSで異常を検出してそれをサンガー法で確認するという作業がどうしても必要になっています。
この先、もっとNGSが進んでいけばこれらの問題も解決できるかもしれませんが、なかなか一筋縄ではいかないようです。
異なるシーケンス技術を使用した多くの異なるNGSプラットフォームがあり、NGSといっても実は非常に多様性に富む表現なのです。しかし、すべてのNGSプラットフォームは、何百万もの小さなDNA断片のシーケンスを並列に実行するという点で共通しています。バイオインフォマティクス解析は、個々のリードをヒトの参照ゲノムにマッピングすることで、これらの断片をつなぎ合わせるために使用されます。ヒトゲノムの30億塩基のそれぞれが複数回配列決定されるため、正確なデータを提供し、予期せぬDNAの変異についての洞察を得ることができます。NGSは、ゲノム全体をシーケンスするために使用することも、22,000個のコード遺伝子(エクソーム全体)または少数の個々の遺伝子を含む、関心のある特定の領域に限定するために使用することもできます。
次世代シーケンシング(NGS)は、ハイスループットシーケンシングとしても知られており、最新のさまざまなシーケンシング技術の総称として使用されています。これらの技術は、以前に使用されていたサンガーシークエンシングよりもはるかに迅速かつ低コストでDNAやRNAのシークエンシングを可能にし、ゲノミクスや分子生物学の研究に革命をもたらしました。
この記事の著者:仲田洋美医師
医籍登録番号 第371210号
日本内科学会 総合内科専門医 第7900号
日本臨床腫瘍学会 がん薬物療法専門医 第1000001号
臨床遺伝専門医制度委員会認定 臨床遺伝専門医 第755号