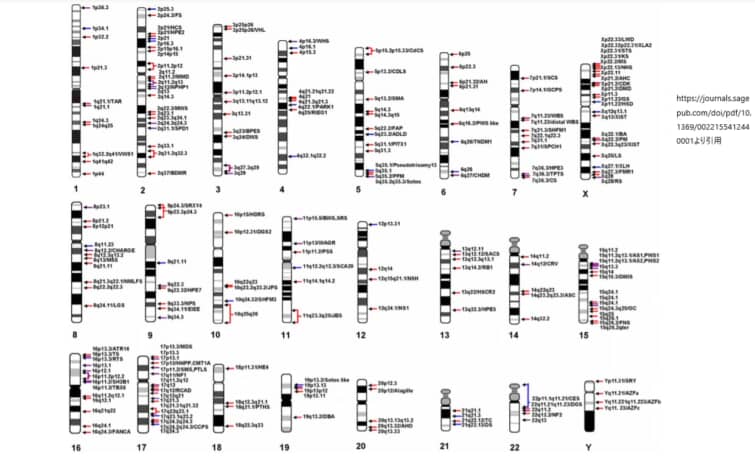
LCR(Low copy repeats)とは、ゲノム内の複数の位置に存在し、高い配列同一性を共有するDNA配列です。これらの繰り返し、またはセグメンタルデュプリケーション(segmental duplications;SDs)は、通常10–300キロベース(kb)の長さであり、95%以上の配列同一性を持ちます[2]。LCRは、ゲノムの不安定性の重要な源であり、ゲノムの変動性と進化を推進すると認識されています[3]。
LCRは、特に霊長類の進化の間に大幅に拡大したため、人間のゲノムの大きな部分を占めています。例えば、人間のY染色体と22番染色体は、それぞれ50.4%と11.9%がSDsで構成されています[2]。LCRのミスアライメント(機械部品やシステムが適切な位置や方向に整列していない状態)は、非アレル同種間相同組換え(NAHR)という重要なメカニズムを介して、染色体のマイクロデリーション(微小欠失)障害およびそれらの相互複製パートナーの基盤となります[2]。
LCRは、ゲノムの進化、適応、および不安定性の推進力としても機能します。二重鎖の切断と修復による連続した転座によって、サブテロメアLCRの構造が形成されることがあります[1]。また、人間のゲノムにおける最大のLCRは、染色体内複製が豊富な間質領域に位置しており、これらの間質LCRのパラログは最も類似性が高いとされています[1]。
LCRは、ゲノムの構造と機能において重要な役割を果たしており、特に22q11.2領域には、ゲノムの他の部分と比較して相対的に多くのLCRが含まれています[5]。これらのLCRは、人間の進化において重要な影響を与えており、特に脳の適応や疾患の発生に関連していることが示唆されています[5]。
LCRの研究は、ゲノムの複雑な領域の解析とアセンブリにおいて依然として課題がありますが、これらの領域の正確なマッピングと特性評価は、進化と疾患の理解に不可欠です[5][6]。
- 参考文献・出典
-
[1] www.explorationpub.com/Journals/em/Article/1001131
[2] en.wikipedia.org/wiki/Low_copy_repeats
[3] www.ncbi.nlm.nih.gov/pmc/articles/PMC6724673/
[4] pubmed.ncbi.nlm.nih.gov/17124407/
[5] www.frontiersin.org/journals/genetics/articles/10.3389/fgene.2021.706641/full
[6] genome.cshlp.org/content/29/9/1389.full
LCRのミスアライメントのホットスポットとは
LCRのミスアライメントのホットスポットとは、LCR(Low Copy Repeats)が集中して存在するゲノム領域で、これらの領域は非対立遺伝子相同組換え(NAHR)の基質として機能し、組換えイベントが頻繁に発生するため「ホットスポット」と呼ばれます. LCRはゲノムの特定の領域に集中しており、これらの領域は遺伝的変異や疾患の原因となる構造的変異を引き起こす可能性が高いです。ミスアライメントは、これらのLCR間での誤ったアライメントを指し、それによってNAHRが誘発されることがあります.
このプロセスは、特定の遺伝的疾患の発生に関連しており、例えば、17p11-12領域のようなホットスポットでは、LCR配列がその領域の大部分を占め、NAHRおよび非相同末端接合(NHEJ)が活発に行われることが知られています. このようなホットスポットは、ゲノムの不安定性を高め、多くの遺伝的疾患のリスクを増加させる要因となります。
非対立遺伝子相同組換え(NAHR)におけるLCRのミスアライメントは、染色体微小欠失疾患および微小重複の根底にある重要なメカニズムとなっています。多くのLCRは、17p11-12領域のような「ホットスポット」に集中しており、その27%はLCR配列で構成されています。この領域内のNAHRおよび非相同末端接合(NHEJ)は、シャルコー・マリー・トゥース症候群1A型、スミス・マジェニス症候群、ポトッキ・ルプスキー症候群を含む幅広い疾患の原因となっています。
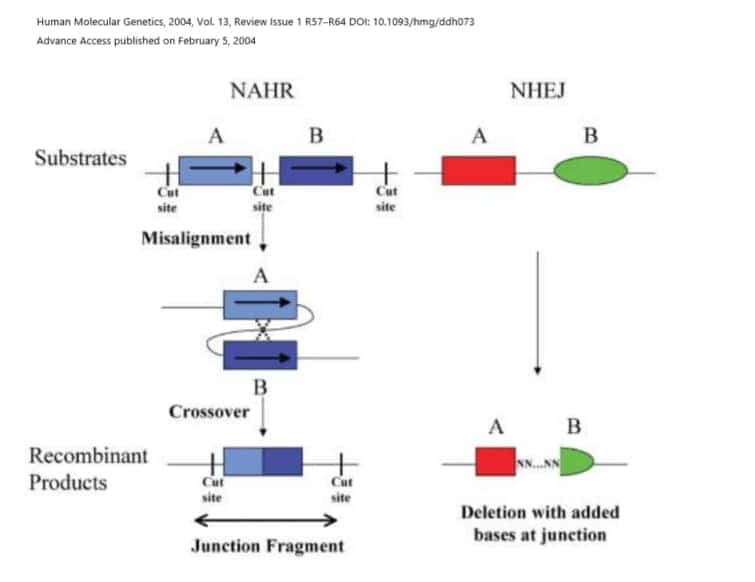
詳しくはNAHRのページをご覧ください。
このようなLCRが多くてCNVがおこりやすい場所がゲノムには数多く特定されています。
Low copy repeat (LCR)の検出方法
Low copy repeat (LCR)の検出方法には、ゲノムの配列解析や特定のDNA断片のコピー数を調べる手法が含まれます。LCRは、ゲノム内で比較的少ないコピー数を持ち、90%以上の相同性を持つ1kb以上の長さの配列ブロックです[8][14]. LCRの検出には、次のような手法が一般的に用いられます:
1. マイクロアレイベースの手法:
– DNAマイクロアレイは、特定のゲノム領域のコピー数変動(CNV)を検出するために使用されます。LCRはCNVの原因となることがあり、マイクロアレイを使用してこれらの変動を検出することができます[4].
2. ポリメラーゼ連鎖反応(PCR):
– PCRは、特定のDNA領域を選択的に増幅し、その存在や量を検出するために使用されます。LCRを含む特定の領域をターゲットにしたPCRは、LCRの存在を確認するために利用されることがあります[15].
3. フルオレッセンス・イン・シチュ・ハイブリダイゼーション(FISH):
– FISHは、染色体上の特定のDNA断片を視覚化するために使用される技術です。LCRを含む領域に特異的なプローブを用いて、LCRの位置やコピー数を検出することができます[7].
4. 次世代シーケンシング(NGS):
– NGSは、ゲノム全体または特定の領域の配列を高速かつ大規模に解析する技術です。LCRを含む領域の配列を詳細に解析し、その構造や変動を検出するために使用されます[16].
5. 比較ゲノムハイブリダイゼーション(CGH):
– CGHは、テストDNAと正常な参照DNAの相対的なコピー数を比較することで、ゲノム全体のコピー数変動を検出する手法です。LCRを含む領域のコピー数変動を検出するために用いられることがあります[5].
これらの手法は、LCRの検出だけでなく、LCRに関連する疾患やゲノムの構造変異の研究にも広く応用されています。また、LCRの検出は、遺伝的疾患の診断や遺伝カウンセリングにおいて重要な情報を提供することができます。
- 参考文献・出典
-
[4] www.tenshi.or.jp/information/other/pdf_genetics/iden_seminar2022_05.pdf
[5] www.jstage.jst.go.jp/article/jmor/30/4/30_145/_pdf/-char/ja
[6] pubmed.ncbi.nlm.nih.gov/7580889/
[7] www.jstage.jst.go.jp/article/ojjscn/46/2/46_131/_pdf
[8] bsd.neuroinf.jp/wiki/%E3%82%B3%E3%83%94%E3%83%BC%E6%95%B0%E5%A4%89%E5%8C%96
[9] jp.rs-online.com/web/content/discovery/ideas-and-advice/lcr-meter-guide
[10] www.jemima.or.jp/tech/3-09-02.html
[11] www.nfcorp.co.jp/techinfo/keisoku/lcr/
[12] www.neb.com/en/faqs/2015/12/13/what-is-ldr-and-how-does-it-differ-from-lcr
[13] edn.itmedia.co.jp/edn/spv/2012/14/news012_6.html
[14] www.hiro-clinic.or.jp/nipt/10q22-3q23-2/