目次
MeRIPseq(MeRIP-seq)とは、Methylated RNA immunoprecipitation sequencingの略で、DNAが転写されてRNAになった後のRNA修飾を検出するための手法です。m6A-seqとも呼ばれています。
DNAのシトシンメチル化やヒドロキシメチル化がゲノムの重要なエピジェネティックな制御因子であるのと同様に、RNAのアデノシン(アデニンとリボースからなるヌクレオシド)メチル化は可逆的な修飾であり、さまざまな生物学的経路や生理学的プロセスに影響を及ぼす可能性が高いことが示されています。
m6Aを含む7,676個の哺乳類遺伝子のmRNAが同定され、m6AがmRNAの共通の塩基修飾であることを示されています。また、m6A修飾は、組織特異的な制御を示し、脳の発達過程で顕著に増加することがわかっています。
m6Aとは?
アデノシンのN(6)位のメチル化(m(6)A)は、RNAの転写後修飾であり、その頻度や生理的な関連性はよくわかっていませんでした。
近年、肥満リスク遺伝子である fat mass and obesity-associated gene; FTO遺伝子がm(6)Aの脱メチル化酵素をコードしていることが判明し、FTOが天然に存在するアデノシンの修飾であるN6-メチルアデノシン(m6A)を脱メチル化する、つまりm(6)AがFTOの生理的プロセスの重要な制御因子であることが示唆されました。
RNA中のm6Aの分布についてはよくわかっていない。m6Aは、tRNA 、rRNA、およびウイルスRNAに存在しています。m6Aは、細胞内RNAの全アデノシン残基の0.1〜0.4%であることが示されており、この修飾がトランスクリプトーム全体に広く存在する可能性が示唆されています。m6Aの存在は何年も前から知られていたのですが、m6Aの存在を検出する方法が確立されていなかったため、mRNA中のm6Aの存在を明らかにすることは、他の修飾塩基に比べて遅れを取ってきました。アデノシンのメチル化は、チミジンやウラシルとの塩基対形成に影響を与えないため、m6Aは標準的なハイブリダイゼーションやシークエンスベースの方法では検出できないためです。
トランスクリプトームにおけるm6Aの頻度、制御、および機能的役割を調べた研究からは、m6Aは発生過程で制御されるRNA修飾であり、動的に変化することがわかってきました。また、m6Aを認識する抗体を用いて、アフィニティエンリッチメント戦略を開発し、次世代シークエンサーと組み合わせることで、m6A部位をハイスループットで同定することがでるようになりました。この手法を用いて、m(6)Aを含む7,676個の哺乳類遺伝子のmRNAを同定し、m(6)AがmRNAの共通の塩基修飾であることを示した。このm(6)A修飾は、組織特異的な制御を示し、脳の発達過程で顕著に増加することがわかった。また、m(6)A部位は、可逆的で広範囲に及ぶ修飾であり、主に進化的に保存された領域に存在し、停止コドンの近傍や3′UTRに集中しており、3’UTR内のm(6)A残基とマイクロRNA結合部位との関連性も明らかになった。これらの知見は、アデノシンメチル化の基質となる転写産物を同定するためのリソースを提供するとともに、哺乳類のトランスクリプトームのエピジェネティックな制御に関する知見を提供するものです。また、m6Aは、停止コドンの近くや3’UTRに非常に多く存在することもわかりました。
m6Aはどこにあるのか?
m6Aの局在化に関する多くの特徴が、ヒトとマウスのトランスクリプトーム間で保存されていること、そしてm6AとmiRNAシグナルの間にこれまで知られていなかった関連性があることがわかりました。これらの研究を総合すると、m6AはmRNAに広く存在し、動的に制御されている塩基修飾であることが明らかになり、m6Aレベルに影響を与えるシグナル伝達経路の影響を受ける可能性の高いmRNAが特定されました。
3’UTR
m6Aの局在化で最も注目すべき点の1つは、3’UTR(3′ 非翻訳領域は成熟mRNA のコーディング領域の下流にあるタンパク質に翻訳されない領域)内での局在化です。3’UTRは、RNAの安定性、細胞内局在性、翻訳制御に影響を及ぼす可能性があるため、RNA制御にとって重要な領域で、これらのイベントのいくつかは、3’UTR内のシス作用のある構造モチーフやコンセンサス配列に結合し、RNAのプロセシングを調整する役割を果たすRNA結合タンパク質(RBP)によって制御されています。m6Aは、メチル化されたDNAのシトシン残基にメチルCPG結合タンパク質2(MeCP2)が結合するのと同様に、特定のRBPの標的mRNAに対する親和性に影響を与えていると考えられます。トランスクリプトーム中にm6Aが豊富に存在し、その局在が広範囲に及ぶことを考えると、m6Aがこのような役割を果たすことは、多くのmRNAの制御に重要な影響を及ぼすと考えられています。
HEK293T細胞でm6Aのプロファイリングを行ったところ、マウスの脳でもメチル化されている何千もの転写産物が見つかった。多くの場合、これらの転写産物におけるm6Aの局在パターンはほぼ同じであることから、一部のRNAは高度に保存されたメチル化プロファイルを持っていることが示唆された。しかし、細胞種ごとに異なるメチル化パターンを示す転写産物も多数発見されたことから、m6Aはそれぞれの細胞環境で異なる制御を受けていることが明らかになった。
また、m6Aのピークを含む3’UTRの大部分にmiRNA結合部位が存在するという結果は、m6AとmiRNAの機能との関連性を強く示唆している。さらに、3’UTR内のm6AピークとmiRNA結合部位は逆の位置にあり、m6A部位は通常、3’UTR内のmiRNA部位と重ならずに先行していることも明らかになった。miRNAは、転写産物の分解または翻訳の抑制のいずれかを促進することで標的mRNAを阻害することができるが(Guoら、2010年、Hendricksonら、2009年)、どちらの運命が優勢であるかを決定する要因はよくわかっていない。考えられるのは、m6AがmiRNA結合部位に近接しているかどうかが、miRNAによる転写抑制のメカニズムに影響を与えることだ。さらに、miRNAの結合が3’UTR内のm6Aレベルに影響を与える可能性もある。実際、発現量の多いmiRNAが発現量の少ないmiRNAよりも有意にm6Aピークに濃縮されているという結果から、miRNAがメチル化状態を制御している可能性が考えられる。
停止コドン付近
また、m6Aは停止コドンの近くで非常に濃縮されています。このように転写産物の中に繰り返し局在することから、停止コドン付近でのアデノシンのメチル化が機能的に重要である可能性を示唆するものです。興味深いことに、アデノシンメチル化のコンセンサスは比較的短く、このコンセンサスに一致する配列はトランスクリプトームの至る所で見られる。しかし、m6Aのコンセンサス部位(特定の機能をもつDNAやRNA領域にみられる特定の塩基配列)が頻繁に存在するにもかかわらず、メチル化は主に停止コドンの近くで起こります。
m6A局在からの今後の課題
このような特異性がどのようにして得られるのか、また、アデノシンメチル化に特異性を与えるために、停止コドンや3’UTRの始まりを認識する細胞メカニズムが関与しているのかどうかを明らかにすることが重要となります。
MeRIP-Seqでトランスクリプトーム全体のm6Aを含むRNAを同定
m6Aの潜在的な役割を理解するためにはトランスクリプトーム全体におけるm6Aの分布を明らかにする必要があります。
6A特異的メチル化RNA免疫沈降法(MeRIP)
m6A抗体を用いてm6Aを含むRNAを濃縮できるかどうかが調べられ、In vitro免疫沈降実験では、m6Aを含む標的に対して、MeRIPを1回行うと約70倍、2回行うと約130倍の濃縮効果が得られることがわかりました。。
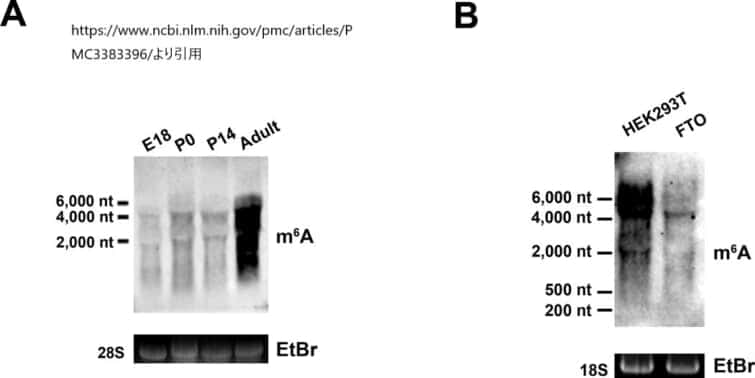
細胞内および発生過程におけるm6Aレベルの制御
A. 脳の発達段階におけるm6Aの存在量の変化。胎生期18日目(E18)、生後0日目(P0)、生後14日目(P14)、成体期のマウスの脳からトータルRNAを単離し、イムノブロット解析を行ってm6Aを含む転写産物を検出した。ローディングコントロールとして、28S rRNAバンドのエチジウムブロマイド染色を示した。
B. FTOは広範な細胞内転写産物を脱メチル化する。FTOをHEK293T細胞で48時間発現させ、細胞内のRNAをイムノブロット分析してm6Aを検出した。
MeRIP-Seq
トランスクリプトーム全体のm6A部位を同定するために、m6A特異的メチル化RNA免疫沈降法(MeRIP)と次世代シーケンサー(RNA-Seq)を組み合わせた方法が開発されました。
MeRIP-Seqの手順
免疫沈降の前に、RNAを約100ntの大きさにランダムに断片化する。m6Aサイトは、免疫沈降した100ntの断片の長さに沿ってどこにでも存在する可能性があるため、シーケンスリードは、m6Aサイトをその中心付近に含む領域にマッピングされると予想されます。極端に言えば、この領域はおよそ200ntの幅があると予測されます(m6Aサイトから上下に100nt)。
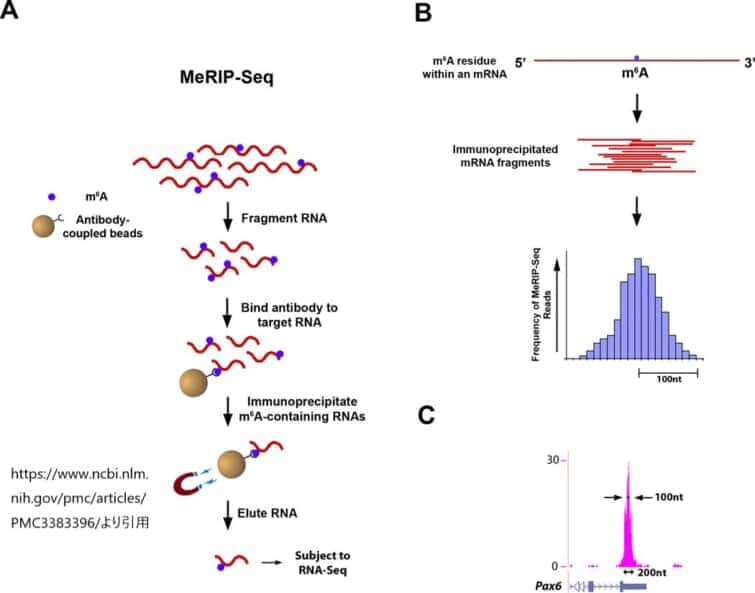
A. MeRIP-Seqの模式図。トータルRNAをRiboMinus処理してrRNA種を除去する。次に、m6Aを含むRNAを、m6A抗体を結合したDynabeadsと混合して免疫沈降させる。その結果、m6Aを含むRNAが高度に濃縮されたRNAプールが得られ、次世代シーケンサーにかけられる。
B. シーケンシングリードと、m6Aサイトを囲むゲノム上の位置とのアライメントの模式図。上:長さ方向に1つのm6A残基を含むmRNA。中:m6A免疫沈降法により単離された100nt幅のmRNA断片で、それぞれが上記のmRNAと同じm6A残基を含む。下:個々の免疫沈降断片の配列を決定して得られたMeRIP-Seqリードの予測頻度を示すヒストグラム。リードの頻度は、m6A部位に近いほど高くなることが予測され、基部で約200nt、中間部で約100ntの幅を持つ「ピーク」を形成する。
C. MeRIP-Seqからのシーケンスリードは、m6Aサイトに収束する。MeRIP-SeqデータからのUCSC Genome Browserの代表的なプロットで、m6A部位(ここではPax6の3’UTRを示す)を囲む典型的なリード頻度のピーク形成を示している。ピークの高さは、マップされた100万リードあたりの塩基あたりのリード数(BPM)で表示される。






