目次
MeDIP mDIP メチル化DNA免疫沈降法
メチル化DNA免疫沈降法(Methylated DNA immunoprecipitation; MeDIPまたはmDIP)は、分子生物学における大規模(染色体またはゲノム全体)な精製技術であり、メチル化DNA配列を濃縮するために使用されます。これは、5-メチルシトシン(5mC)に対する抗体を介してメチル化されたDNA断片を分離するものである。この技術は2005年にWeberらによって初めて発表され、高解像度DNAマイクロアレイ(MeDIP-chip)や次世代シーケンシング(MeDIP-seq)などのハイスループットなDNA検出法に精製されたメチル化DNA断片を入力することができるためメチローム(DNAのメチル化パターン)レベルの評価が可能となった。しかし、メチロームの理解はまだ初歩的なものであり、他のエピジェネティックな特性と同様に、パターンが細胞の種類によって異なるという事実によって、その研究は複雑なものとなっている。
DNAのメチル化とは
DNAのメチル化は、メチル基転移酵素によるシトシンの5位の可逆的なメチル化を指し、多細胞生物における主要なエピジェネティックな修飾である。 哺乳類では、この修飾は主にCpGサイトで起こり、CpGアイランドと呼ばれる領域に集まりやすい。CpGアイランドの中には、転写開始点のプロモーター領域と重なったり、近接したりするものがわずかに存在する。また、他の部位でも修飾が起こる可能性があるが、いずれの部位のメチル化も、転写因子の結合を阻害するか、クロマチン構造を抑制的な状態に変化させることで、遺伝子発現を抑制する。
DNAメチル化の役割を理解は、疾患の研究のなかで大きく進んできた。現在、主な研究テーマは、がんなどの疾患を調査して、メチル化が大きく変化したDNA領域を特定することである。これらの領域に含まれる遺伝子は、病気の根本的な遺伝的原因を説明できる可能性があるため、機能的にも興味深い。例えば、がん細胞の異常なメチル化パターンは、当初、がん抑制遺伝子が沈黙するメカニズムとして示されていたが、その後、より広範な種類の遺伝子が影響を受けることが確認されてきた。
メチル化解析のその他の技術
メチル化解析には、タイピング技術とプロファイリング技術の2つのアプローチがある。
タイピング技術は、多数のサンプルの中から少数の遺伝子座を対象とし、PCR、制限酵素、質量分析などの技術を用いて行う。MeDIPのようなプロファイリング技術は、ゲノムまたはメチローム全体のメチル化を評価することを目的としており、制限酵素を用いたゲノムスキャン(RLGS)や、DNAを重亜硫酸塩で処理してメチル化されていないシトシン残基をウラシルに変換する重亜硫酸塩変換法などがある。
メチル化解析のその他の技術の限界
メチルロームのマッピングやプロファイリングを行う他の方法は有効であるが、分解能、スループットのレベル、実験のバリエーションに影響を与える制限がないわけではない。例えば、RLGSでは、制限酵素の標的となるゲノム中の制限部位の数に制限があり、通常、評価できるランドマークの数は最大で約4100個である。重亜硫酸塩配列に基づく方法は、1塩基の分解能が可能であるにもかかわらず、メチル化されていないシトシンをウラシルに変換する際に不安定になるという欠点がある。全ゲノムを網羅的にプロファイリングできるマイクロアレイは、ユニークなプローブが少なくなるため、設計が難しくなる。
MeDIPの方法
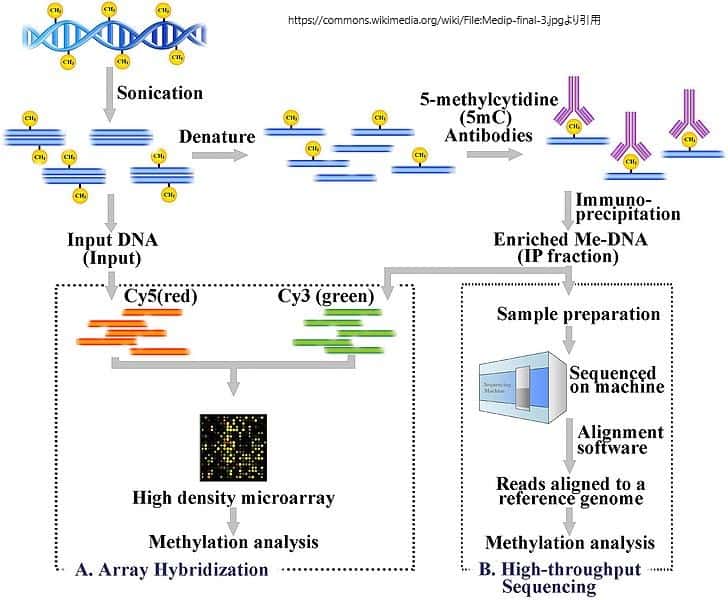
MeDIPには高解像度アレイハイブリダイゼーションまたはハイスループットシーケンシングを組み合わせた方法がある。それぞれのDNA検出方法について、実験後の処理や解析についても簡単に説明する。メチル化された配列を同定するために使用した技術に応じて、生データの異なる後処理が必要となる。これは、ChIP-chipやChIP-seqを用いて生成されたデータと類似している。
MeDIP手順のワークフロー概要
MeDIPの後にアレイハイブリダイゼーション(A)またはハイスループット/次世代シーケンシング(B)を行う。
1.メチル化DNA免疫沈降法(MeDIP):共通過程
細胞からゲノムDNAを抽出(DNA抽出)し、精製する。精製したDNAに超音波をかけて、ランダムな断片にせん断する。この超音波処理は、迅速かつ簡単で、制限酵素の偏りを避けることができる。得られる断片の長さは300~1000塩基対(bp)であるが、一般的には300~600bpである(文献)。これらの断片の長さが短いことは,十分な分解能を得ること,免疫沈降の下流工程の効率を高めること,断片の長さによる影響やバイアスを減らすことに重要となる。また,5-メチルシチジン(5mC)抗体の結合には,1本の5-methyl-cytidine (5mC)だけではなく,複数の5mCが必要となるため、断片の大きさは5-メチルシチジン(5mC)抗体の結合に影響を与える。
変性後、このDNAをモノクローナル5mC抗体とインキュベートする。
次に、古典的な免疫沈降法を適用する。抗マウスIgGを結合させた磁気ビーズを用いて抗5mC抗体を結合させ、結合していないDNAを上澄み液中に除去する。
DNAを精製するために、プロテイナーゼKを加えて抗体を消化してDNAを放出させ、これを回収してDNA検出の準備をする。
2.MeDIPとアレイベースのハイブリダイゼーション(MeDIP-chip)
超音波処理後に得られた入力DNAの一部をシアニン-5(Cy5;赤)デオキシ-シトシン-三リン酸で標識し、免疫沈降ステップ後に濃縮されたメチル化DNAをシアニン-3(Cy3;緑)で標識する。標識されたDNAサンプルは、2チャンネルの高密度ゲノムマイクロアレイ上で一緒にハイブリットし、存在量と相対量をプローブする。この比較の目的は、ハイブリダイゼーションレベルに有意な差を示す配列を特定することで、目的の配列が濃縮されていることを確認することである。アレイを用いたMeDIP配列の同定は、アレイのデザインに制限がある。結果として、分解能はアレイデザインのプローブに限定される。ほとんどのアレイ技術と同様に、ノイズなどのハイブリダイゼーションの問題を補正するために、信号処理に必要な標準的なステップが追加される。
3.MeDIPとハイスループットシーケンシング(MeDIP-seq)
MeDIP-seqは、MeDIPと454 pyrosequencingやIllumina (Solexa)などの次世代ショートリードシーケンシング技術を組み合わせたものである。このショートリードは、Mapping and Assembly with Quality(Maq)などのアラインメントソフトウェアを用いて参照ゲノムにアラインメントされる。
データの品質と精度を評価するアプローチの検証は、定量的PCRで行うことができる。これは、MeDIPサンプルの配列をメチル化されていないコントロール配列と比較することで行われる。そして、サンプルをゲル上で走らせ、バンドの強度を比較する。相対的な強度は、エンリッチメントを見つけるためのガイドとなる。また、この結果をMeDIP-chipの結果と比較することで、必要なカバレッジを判断することができます。
ダウンストリームバイオインフォマティクス解析
MeDIP-seqを用いた典型的な実験を示すシンプルなワークフローに必要な過程である。
MeDIPによって生成されたデータを観察すると、ゲノム上のメチル化されたCpGサイトの密度が異なるために、DNAメチル化レベルの推定値が混乱することがある。これは、CpG-poor(低密度)な領域を解析する際に問題となることがある。この密度の問題の理由の一つは、免疫沈降の効率に影響を与えることである。これを改善するために、メチル化されたCpGサイトの密度をモデル化することで、MeDIPで生成されたデータから絶対的なメチル化レベルを推定するツールが研究開発された。このツールはBayesian tool for methylation analysis(Batman)と呼ばれている。この研究では、プロモーター、遺伝子コーディング領域、アイランド、制御要素にあるCpGサイトのうち、メチル化レベルを推定できる部分のカバレッジが約90%であることが報告されており、これはこれまでの方法と比較して約20倍のカバレッジとなっている。
MeDIP-seqやMeDIP-chipを用いた研究は、いずれもゲノムワイドなアプローチであり、メチロームの機能マッピングを得るという共通の目的を持っています。DNAメチル化領域が特定されると、様々なバイオインフォマティクス解析を行って、生物学的な疑問に答えることが可能となる。1つの明白なステップは、これらの領域に含まれる遺伝子を調査し、その抑制の機能的意義を調べることである。例えば、がんにおける腫瘍抑制遺伝子のサイレンシングは、DNAメチル化に起因すると考えられています。既知の腫瘍抑制遺伝子のハイパーメチル化とそれに続く抑制を引き起こす突然変異イベントを特定することで、病気の原因となる要因をより具体的に特定することが可能となる。あるいは、正常にメチル化されていることが知られているが、何らかの突然変異現象の結果、もはや抑制されていない遺伝子を特定することもできる。
また、DNAメチルトランスフェラーゼ(DNMT)などのエピジェネティックな制御因子が影響を受けていないかどうかを調べて特定することも可能である。
ハイスループットのメチル化データ(MeDIP-seqやMeDIP-ChIPなど)に遺伝子セット解析(DAVIDやGoSeqなど)を適用すると、大きなバイアスがかかることが指摘されているが、サンプルラベルのパーミュテーションを用いたり、統計モデルを用いて各遺伝子をターゲットとするCpGプローブやCpGサイトの数の違いをコントロールしたりすることで補正することができるとされている(文献)。
MeDIPの限界
MeDIPを使用する際に注意すべき限界は、典型的な実験的要因である。これには、手順に使用する5mC抗体の品質と交差反応性が含まれる。さらに、DNA検出法(アレイハイブリダイゼーションやハイスループットシーケンサーなど)には、一般的に確立された制限があります。特にアレイベースの手順では、上述したように、分析される配列は、使用される特定のアレイデザインに限定される。
ハイスループット、次世代シーケンシングにも最も典型的な限界が存在する。ゲノムの反復領域に対するアラインメント精度の問題は、それらの領域におけるメチル化の分析の精度を低下させる。






